
Exemples de cas et propositions de schémas d’interprétation
Auteurs :
Dr Camille Levesque, chef de service Virologie et Sérologie au LABOCEA 35, camille.levesque@labocea.fr
Dr Guillaume Lequeux, chef de service Bactériologie et parasitologie au LABOCEA 35, guillaume.lequeux@labocea.fr
Dr Thibault Jozan, vétérinaire responsable technique MSD Santé Animale.
Introduction
De nouveaux outils de diagnostic ont été développés et sont proposés par les laboratoires : identifications bactériennes par spectrométrie de masse MALDI-TOF, PCR, séquençage, ou de façon indirecte par méthode ELISA. Cependant, l’interprétation des résultats d’analyse obtenus peut être problématique car la détection des agents peut dépendre du type de prélèvement (Ecouvillon Nasal Profond – ENP, Aspiration TransTrachéale – ATT, Lavage Broncho-Alvéolaire – LBA, Poumon), du nombre d’animaux prélevés, du délai entre l’apparition des premiers symptômes et le prélèvement, de l’immunité des animaux et des limites propres à chaque méthode d’analyse.
De plus, l’implication de certains pathogènes dans les pathologies respiratoires fait toujours l’objet de controverses (1,2). En effet, en dehors des agents primaires reconnus, la détection d’agents dits « secondaires » pose question. Or, ces agents opportunistes peuvent être de simples contaminants, appartenir à la flore commensale de l’appareil respiratoire des animaux sains (3,4,5,6) ou encore se multiplier à la faveur de conditions favorisantes infectieuses ou non, d’une baisse d’immunité et participer à la dégradation de la santé de l’animal.
En complément de l’identification d’agents avec une bonne sensibilité et spécificité, les outils diagnostiques doivent permettre une différenciation entre une contamination (lors de la réalisation du prélèvement par exemple), portage (présence sans inflammation, ou modification de l’état de santé de l’animal) et infection de l’appareil respiratoire avec répercussion subclinique ou clinique. Est-ce que l’association de différentes analyses peut pallier les limites rencontrées à l’heure actuelle dans l’interprétation de certains résultats ?
Les principaux intérêts et limites des analyses de laboratoire les plus courantes seront présentés ainsi que des exemples d’associations d’analyses réalisées à Labocea et d’interprétation des résultats. Enfin, des schémas d’aide pour le choix des analyses à réaliser en fonction de la situation épidémiologique et de l’objectif recherché sont proposés.
Intérêts et limites de la PCR
Lors d’épisode respiratoire, le recours aux analyses PCR est fréquent.
A Labocea, entre 2017 et 2022, près de 1400 analyses de PCR temps-réel multi-agents avec le kit VetMAX™ Ruminant Respiratory Screening Kit (Thermofisher scientific) permettant la mise en évidence du génome de quatre bactéries (Mannheimia haemolytica (Mh), Pasteurella multocida (Pm), Histophilus somni (Hs) et Mycoplasma bovis (Mb)) et trois virus (VRSB, Pi3 et Coronavirus bovin (BCoV)) simultanément, ont été réalisées. Les PCR développées et validées sont en général considérées comme très sensibles (seuil de détection bas et capacité à détecter des bactéries ou virus viables ou non). Cela se traduit par la détection dans une grande majorité des cas d’au moins un agent par analyse réalisée. Ainsi, à Labocea, la proportion d’analyses avec au moins un agent détecté est de 92 % entre janvier 2017 et janvier 2022 avec la PCR multi-agents.
La PCR bénéficie d’une très bonne sensibilité, y compris sur mélange de cinq prélèvements de nature identique. En augmentant le nombre d’animaux prélevés, le risque de faux négatifs par rapport aux agents recherchés est limité (2).
D’après les résultats obtenus à Labocea, à l’exclusion d’Histophilus somni, la fréquence de détection des autres agents recherchés est significativement plus importante (p<0.005) par les analyses de mélanges de deux à cinq écouvillons (tableau 1).
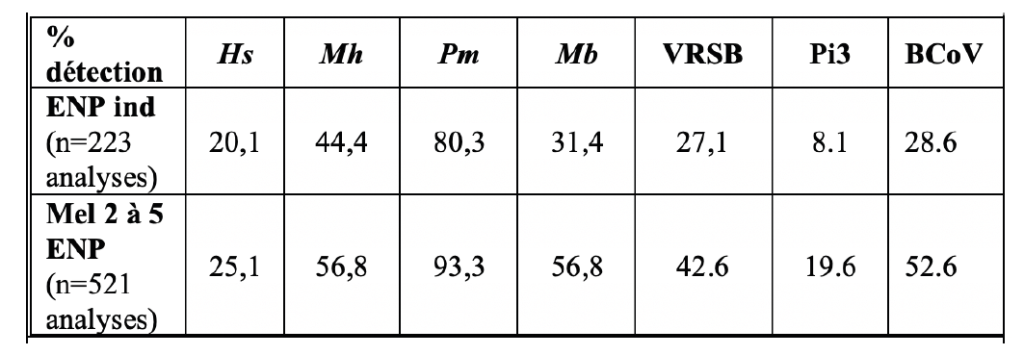
PCR temps-réel multi-agents avec le kit VetMAX™ Ruminant Respiratory Screening Kit (Thermofisher scientific) permettant la mise en évidence du génome de 4 bactéries (Mannheimia haemolytica (Mh), Pasteurella multocida (Pm), Histophilus somni (Hs) et Mycoplasma bovis (Mb)) et 3 virus (VRSB, Pi3 et Coronavirus bovin (BCoV).
La fréquence de détection des différents agents pathogènes peut différer selon la matrice prélevée (1,2,3,7) avec des observations parfois contradictoires. Lowie T. et al. (7) indiquent une meilleure détection du BCoV et du VRSB sur lavage broncho-alvéolaire LBA que sur ENP. Dans une étude de Meyer et al. (10) dans laquelle des ENP et LBA ont été réalisés en parallèle sur les mêmes veaux, le VRSB est détecté 4x plus sur LBA alors que le BCoV est détecté 1,5 fois plus sur ENP.
D’après les résultats obtenus au laboratoire depuis 2017, la fréquence de détection du Coronavirus bovin est significativement différente (p<0.001) sur les prélèvements individuels d’ENP (28.6 %) par rapport aux poumons (10.7 %). De même, Mannheimia haemolytica et Pasteurella multocida ont des fréquences de détection apparentes significativement différentes (p<0.001) entre ENP individuel (Mh : 44 %, Pm 80 %) et poumons (Mh 61 % et Pm 57 %).
Attention, dans les données de Labocea présentées ci-dessus (comparaison selon la matrice et le nombre d’échantillons analysés dans le mélange), s’agissant de prélèvements dans des élevages différents, les différences peuvent être potentiellement le reflet de contextes épidémiologiques cliniques différents, ayant conduit à des choix de prélèvements différents.
Par ailleurs, comme pour toute analyse de laboratoire, le délai entre la date d’apparition des premiers symptômes et le prélèvement est à prendre en compte dans l’interprétation des résultats d’analyses.
Lorsque le vétérinaire intervient, il n’est pas rare que le début de l’épisode remonte déjà à plusieurs jours, diminuant ainsi fortement la valeur prédictive de l’implication ou non des agents recherchés par analyses directes (11) :
- un risque de faux négatifs vis-à-vis d’agents primaires ou initiateurs (2,7).
Les virus et certaines souches bactériennes à pouvoir pathogène ou de diffusion élevé, comme démontré pour M. haemolytica et M. bovis (8,9) ont un rôle primaire important lors d’épisode respiratoire d’allure épidémique. Les infections virales sont sous-détectées lorsque les prélèvements ne peuvent pas être réalisées en début d’infection (2,7).
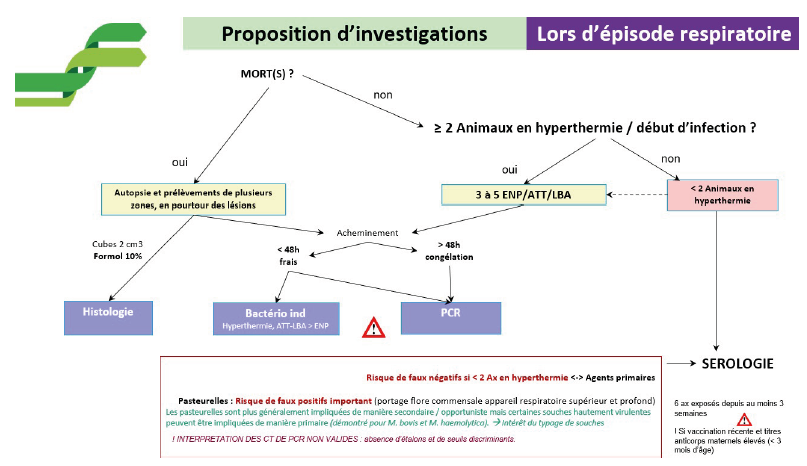
Lors de mortalité, il n’est pas rare de retrouver uniquement des pasteurelles sur des PCR de poumon alors que l’intervention d’un virus est démontrée par la réalisation d’analyses complémentaires comme l’histologie et/ou des sérologies sur les congénères (voir III et IV).
Il paraît donc important de prélever plusieurs animaux en début d’infection afin de limiter les risques de ne pas détecter les agents primaires ou initiateurs (2,7,8,9).
La connaissance de traitements ou de vaccination antérieures est également à prendre en compte dans l’interprétation des résultats. En effet, il existe également :
- un risque de faux négatifs vis-à-vis des bactéries lors de traitement antibiotique, même si ce risque est beaucoup plus limité qu’en culture bactériologique,
- un risque de faux positifs pour VRS et Pi3 lors de vaccination avec des agents vivants atténués en intranasal. Des faux positifs vis-à-vis de ces agents sont possibles par les méthodes PCR jusqu’à 1 mois après la vaccination pour VRSB (12).
NB : La détection des agents couverts par un vaccin ne suffit pas en elle-même à caractériser une immunodéficience ou un échec vaccinal.
Les différents vaccins respiratoires du marché peuvent obtenir des indications de réduction de l’excrétion, de la clinique, voire des lésions propres à chaque vaccin et chaque valence. La vaccination vise donc à réduire les conséquences d’une infection vis-à-vis des principaux agents représentant le plus grand risque. Il n’est pas anormal de retrouver par PCR des agents ciblés par le protocole vaccinal chez les animaux correctement immunisés, en bonne santé.
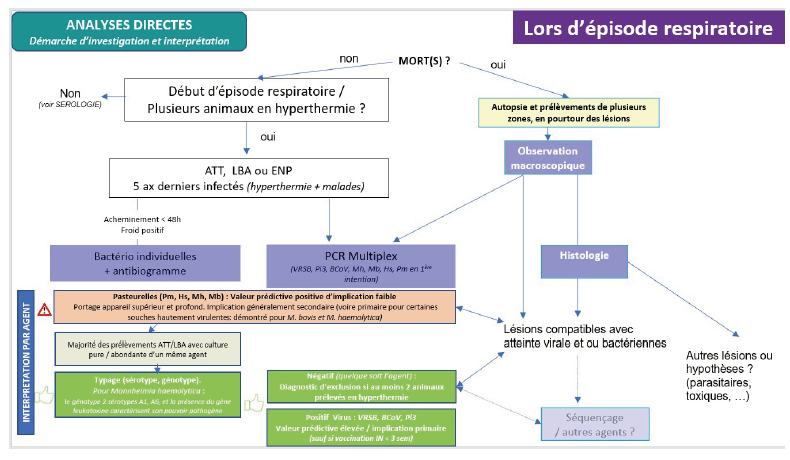
Enfin, l’interprétation vis-à-vis des pasteurelles reste délicate :
la détection par PCR de Hs, Mh, Pm ou même Mb, du fait de leur portage au niveau de la flore de l’appareil respiratoire supérieur et profond pose la question de l’interprétation des résultats positifs pour ces agents (encadré 1).
Encadré 1 : L’appareil respiratoire profond n’est pas stérile !
Quid de l’identification de pasteurelles sur poumon, aspiration transtrachéale (ATT), lavage broncho-alvéolaire (LBA) et écouvillon nasal profond (ENP) en pratique ?
Le temps où l’on pensait que le seul diagnostic de pasteurelles au niveau respiratoire profond permettait de signer leur implication au moins secondaire est révolu.
De nombreuses publications (2,4,5,6,13,14) rapportent la détection fréquente de pasteurelles par PCR ou bactériologie sur des prélèvements ATT ou LBA en l’absence d’épisode respiratoire et/ou sur des animaux « sains » au niveau clinique, échographique et/ou des marqueurs de l’inflammation.
Dans l’étude de Angen O. et al. (5), des aspirations transtrachéales ont été réalisées sur des veaux d’élevages laitiers visités en été en l’absence d’épisode respiratoire, puis en hiver lors d’épisode respiratoire. Des pasteurelles ont été identifiées chez 68 % des veaux prélevés par ATT en l’absence d’épisode respiratoire ; 59 % par culture et 41 % par PCR. Un élevage sur deux avait plus de 30 % d’ATT positifs vis-à-vis de Histophilus somni, 4/6 avec plus de 50 % pour Pasteurella multocida. Pour Mannheimia haemolytica, cinq élevages sur six avaient moins de 20 % d’ATT positifs (tableau 1). En pratique, la réalisation de PCR par pool de trois à cinq prélèvements augmente cette probabilité de détection déjà élevée en l’absence de maladie.
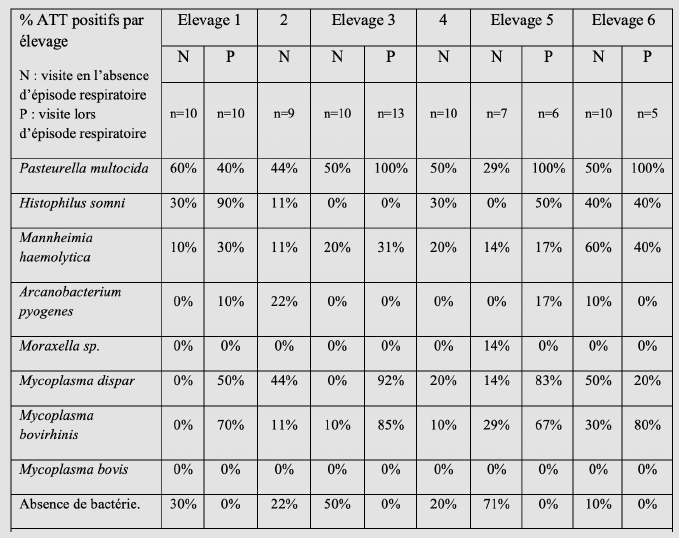
Est-ce qu’une analyse quantitative permettrait de faire la distinction entre la colonisation sans conséquence et la surinfection avec dégradation de l’état de santé ?
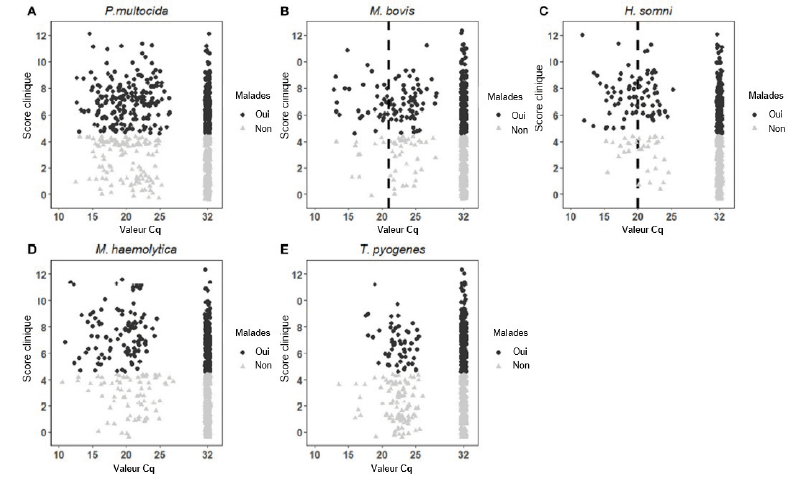
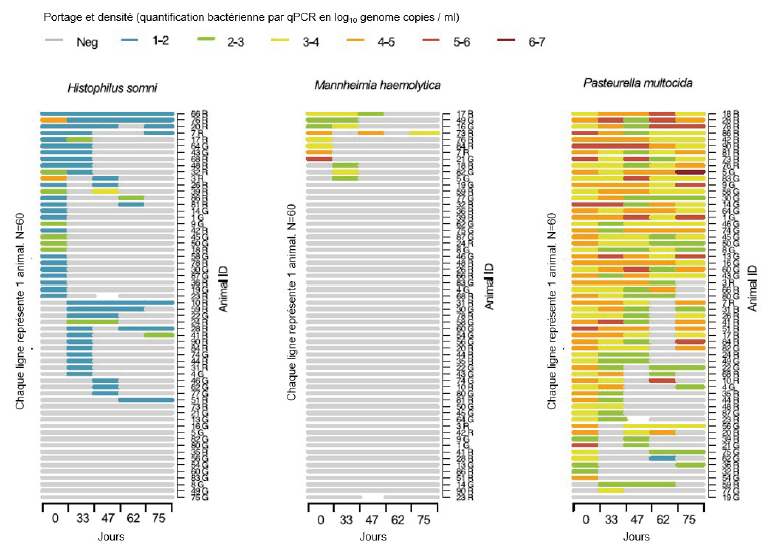
Une étude de Klompmaker et al. (13) en particulier a été menée dans ce sens. Pour H. somni, un CT ≤ 17,4 était associé à 86 % des veaux malades mais un CT > 17,4 était aussi associé à 40 % de veaux malades. L’établissement de seuil ne semble pas suffisamment discriminant dans la mesure où une proportion relativement importante d’animaux classés comme sains pouvait avoir un CT inférieur au seuil choisis, en particulier pour P. multocida et M. bovis. Aucun seuil n’a pu être établi pour T. pyogenes et Mannheimia (figure 1). Les auteurs précisent qu’il conviendrait de distinguer les différents sérotypes de M. haemolytica afin de prendre en compte leurs différences de pouvoir pathogène (encadré 2).
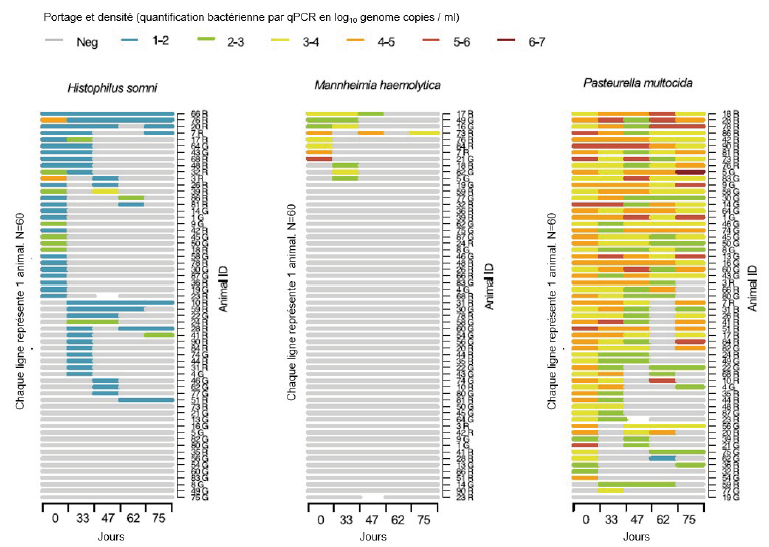
L’établissement de seuil ne semble pas une piste suffisamment aboutie à ce jour, ce qui est corroboré aussi par l’étude de Thomas et al. (4). Cette étude présente les valeurs obtenues en PCR quantitatives de portages asymptomatiques des Pasteurellaceae sur des écouvillons de veaux allaitants sevrés, cliniquement sains sur une durée de 75 jours. Ces veaux avaient été allotés 2 mois avant le début des prélèvements et n’ont pas été déplacés. 95 % des veaux étaient plusieurs fois positifs pour P. multocida, 75 % pour H. somni et 13 % pour M. haemolytica. La distribution des densités bactériennes mesurées par qPCR était très étendue (figure 2).
À ce jour, aucune donnée et méthode ne permettent d’établir de lien fiable et valorisable en pratique entre le CT et l’imputabilité de l’agent détecté.
Une des dernières limites de la PCR est qu’on ne peut détecter que ce que l’on recherche a priori et en fonction des kits disponibles. D’autres agents tels que le BCoV ou l’influenza D ne font pas toujours partie des kits utilisés en routine en première intention alors que leur implication dans le complexe respiratoire bovin parait fréquente et est de plus en plus reconnue (2,10).
Au vu des limites de la PCR, il apparaît alors pertinent d’associer la PCR à d’autres méthodes d’analyses (séquençage, bactériologie, MALDI-TOF, sérologie). Des exemples d’associations de la PCR avec d’autres méthodes sont présentés ci-après (encadrés 2 et 3).
Intérêts et limites de la bactériologie
L’interprétation de la bactériologie se fait en tenant compte de différents facteurs, comme pour la PCR (conditions de prélèvements et échantillonnage, matrice, traitements antibiotiques éventuels).
Il est important de tenir compte :
- des commémoratifs cliniques et épidémiologiques et d’investigation des facteurs de risques qui permettent de fortement orienter vers une étiologie plutôt qu’une autre (7),
- du potentiel de chaque agent d’agir comme pathogène primaire ou secondaire par rapport à son portage au niveau de la sphère respiratoire supérieure et profonde (1,2).
La prise en compte des éléments de pureté et d’abondance des colonies est capitale à l’interprétation pour toutes les bactéries.
On pourrait penser que l’identification sur plusieurs prélèvements ATT/LBA d’animaux en début d’infection, d’une même pasteurelle en culture pure et abondante présage de son implication au moins secondaire. Il convient toutefois de rester prudent. Van Leenen et al. (3) n’ont pas mis en évidence de différence de taux d’isolement bactérien et des résultats quantitatifs entre les cultures bactériologiques réalisées sur les LBA de veaux sains et ceux diagnostiqués en infection respiratoire subclinique ou clinique.
En complément de la culture bactériologique, la recherche de facteur de virulence par PCR ou MALDI-TOF ou séquençage permet de différencier des souches selon leur potentiel pathogène ou de résistance. Ces investigations complémentaires se déploient en laboratoire d’analyse et permettent d’apporter de nouvelles données intéressantes pour interpréter les résultats. Pour Mannheimia haemolytica, il est maintenant possible de déterminer le génotype et sérotype d’une souche ainsi que la présence ou non du gène de la leucotoxine. Ces éléments informatifs sont intéressants pour interpréter les résultats (encadré 2). Dans une étude réalisée chez des jeunes bovins à l’engraissement en Pays de la Loire, 63 % des souches isolées correspondaient à du sérotype A6. Les sérotypes A1 et A2 ne représentaient que 30 % et 7 % des cas respectivement (8).
Encadré 2 : Quelles informations apporte la détermination du génotype et du sérotype des souches de M. haemolytica isolées en pathologie respiratoire ?
Les outils diagnostiques permettant une caractérisation plus fine des souches de M. haemolytica impliquées en pathologie respiratoire bovine sont à présent relativement facilement accessibles en routine :
- la détermination du génotype est permise lors de l’identification des souches par spectrométrie de masse MALDI-TOF, méthode d’identification qui se généralise et devient courante en diagnostic vétérinaire (20),
- la détermination du sérotype peut être faite par PCR temps-réel, méthode qui s’est substituée au sérotypage par agglutination (21). Les PCR temps-réel actuellement disponibles permettent également de détecter sur les souches isolées la présence du gène codant pour la leucotoxine (LTx), principal facteur de virulence. La détection de ce gène sur une souche isolée en contexte clinique et/ou lésionnel permet de lui attribuer avec une forte probabilité l’origine des symptômes et/ou lésions observées.
Une étude américaine récente concernant Mannheimia haemolytica a permis de montrer que seules les souches appartenant au génotype 2 peuvent être considérées comme pathogènes (car systématiquement retrouvées dans les poumons d’animaux malades, au contraire du génotype 1 qui correspond aux souches commensales retrouvées exclusivement dans le nasopharynx d’animaux sains). Par ailleurs, les supports de résistance voire de multirésistance aux antibiotiques (intégrons) sont exclusivement retrouvés chez les souches de génotype 2 (20).
Des auteurs ont réussi à déterminer qu’il existe des pics spécifiques à chacun de ces deux génotypes en spectrométrie de masse MALDI-TOF, permettant ainsi de les différencier de façon rapide, sans nécessité d’effectuer un génotypage complet (20). Ces travaux ouvrent la possibilité pour le praticien de connaître dès le lendemain de la réception d’un échantillon au laboratoire le génotype d’une souche de M. haemolytica qui serait isolée et donc son pouvoir pathogène mais également son profil de résistance probables.
Dans la séquence pathogénique des infections à M. haemolytica, il a été démontré que les bovins sains sont le plus souvent colonisés par des souches commensales de sérotype A2, mais que les infections respiratoires sont le plus fréquemment causées par des souches pathogènes appartenant au sérotype A1 (22). Les raisons de cette transition de populations bactériennes majoritairement de sérotype A2 vers une « explosion » de populations bactériennes de sérotype A1 dans le tractus respiratoire ne sont pas complètement connues, mais il semblerait que seules les souches de sérotype A1 possèdent des facteurs de virulence permettant l’invasion puis leur prolifération à l’intérieur des cellules de l’épithélium respiratoire (22).
Aussi l’avènement de l’identification bactérienne par MALDI-TOF a permis de mettre en évidence de façon plus simple, fiable et rapide, d’autres pasteurelles potentiellement impliquées en pathologies bovines.
Deux espèces appartenant à la famille des Pasteurellaceae ont notamment fait l’objet de descriptions récentes (< 10 ans) en tant qu’agents impliqués en pathologie bovine : Bibersteinia trehalosi et Gallibacterium anatis.
Bibersteinia trehalosi est une bactérie principalement décrite comme agent responsable de septicémies aigües (« pasteurellose systémique ») chez les agneaux de plus de 6 mois. Cette espèce avait déjà été décrite comme commensal possible du nasopharynx, des conjonctives oculaires et des amygdales chez les bovins (23).
Chez les bovins, différentes présentations cliniques lors d’infections à Bibersteinia trehalosi sont décrites particulièrement depuis une dizaine d’années : hépatite nécrosante et sérosite fibrineuse chez des vaches adultes, bronchopneumonies fibrino-purulentes voire nécrosantes chez des bovins de plus de mois (24). Des cas de pleuropneumonies associées à des septicémies ont également récemment été décrits chez des veaux laitiers nouveau-nés (25).
Bibersteinia trehalosi est à présent reconnu en Amérique du Nord comme agent bactérien émergent, pathogène primaire impliqué dans le complexe respiratoire bovin chez les bovins en engraissement mais aussi chez de jeunes veaux (26). Cet agent est de plus en plus isolé dans ces contextes cliniques depuis 2007 environ.
Il est possible, selon les auteurs de ces différentes études, que l’implication de Bibersteinia trehalosi en pathologie bovine ait été sous-estimée pendant des années du fait des difficultés existantes à la différencier de M. haemolytica lors de son identification bactériologique, alors que les tableaux lésionnels liés à ces deux bactéries ne sont pas différenciables. L’avènement de l’identification bactérienne par spectrométrie de masse MALDI-TOF, permettant une identification facile et fiable de cette espèce bactérienne pourrait être une explication de l’augmentation de son incidence dans l’espèce bovine.
Il est intéressant de noter que M. haemolytica et Bibersteinia trehalosi partagent des facteurs de virulence communs (lipopolysaccharides et leucotoxine) et que l’utilisation de vaccins dirigés contre M. haemolytica a démontré une protection croisée contre les infections à Bibersteinia trehalosi chez les bovins (27).
Enfin, les études de séquençage tendent à démontrer que, dans les troupeaux mixtes ovins-bovins, la transmission de cette bactérie se ferait plutôt dans le sens bovins vers ovins que l’inverse (25).
En France, peu de données existent, mais il est constaté un à deux isolements par an depuis 2017 environ à LABOCEA Fougères alors que de tels isolements n’avaient pas été constatés précédemment. Il est néanmoins possible, là encore, que ce constat puisse en partie être lié à l’utilisation en routine du MALDI-TOF pour les identifications bactériennes depuis quelques années.
Gallibacterium anatis a fait l’objet d’une description beaucoup plus récente. Il s’agit d’une bactérie émergente chez les volailles (espèce poule en particulier), pathogène opportuniste responsable principalement de septicémies (salpingites, péritonites, épididymites et lésions respiratoires) dans cette espèce.
Gallibacterium anatis avait précédemment été isolée à partir de fèces chez des bovins sains, mais une récente étude en Belgique suggère de considérer cette bactérie comme pathogène respiratoire émergent, secondaire et opportuniste chez les bovins (28). Dans cette publication, aucun lien n’avait pu être fait avec une contamination éventuelle à partir d’élevages aviaires. Il semble que, comme précédemment décrit pour Bibersteinia trehalosi, l’incidence de Gallibacterium anatis chez les bovins ait augmenté ces dernières années du fait d’une utilisation plus large du MALDI-TOF, outil qui aurait seul ou en grande parti augmenté le taux d’identification de cette bactérie.
La préoccupation majeure, selon ces auteurs (28) concernant Gallibacterium anatis réside dans le constat (partagé par les observations faites à LABOCEA Fougères) de fréquentes multi-résistances chez les souches isolées de pathologies respiratoires bovines, dont des résistances fréquentes aux fluoroquinolones.
Enfin, la bactériologie reste incontournable pour réaliser des antibiogrammes et suivre régulièrement l’évolution des sensibilités des antibiotiques du protocole thérapeutique vis-à-vis des pasteurelles.
Encadré 3 : Association des analyses PCR 7 agents respiratoires et de la bactériologie classique.
Analyses des données comparées des forfaits respiratoires réalisés à Labocea.
53 analyses ont été réalisées en parallèle en bacteriologie et en PCR entre décembre 2020 et février 2022 à partir du même prélèvement de départ. La bactériologie consistait à réaliser une culture bactérienne sur milieux sélectifs et non sélectifs puis à identifier les colonies d’intérêt par MALDI-TOF (à l’exclusion de Mycoplasma bovis).
Pour la PCR, le kit VetMAX™ Ruminant Respiratory Screening Kit (Thermofisher scientific) est utilisé et permet la recherche des 7 agents simultanément : Histophilus somni (H. somni), Mannheimia haemolytica (M. haemolytica), Pasteurella multocida (P. multocida), Mycoplasma bovis (M. bovis), Coronavirus bovin (BCoV), Parainfluenza 3 (Pi3) et le virus respiratoire syncitial bovin (VRSB).
Les prélèvements reçus sont principalement des écouvillons nasaux pharyngés (31 dossiers), puis des poumons (n= 15), ATT (4) et LBA (3). Les demandes d’analyse de mélanges sont majoritaires pour les ENP reçus. La moitié des demandes au moins concernent des bovins âgés entre un et six mois.
Pour 50 analyses sur 53 réalisées au moins un agent microbien potentiellement pathogène a été détecté. De plus, 67 % des prélèvements analysés ont obtenus des résultats concordants entre la bactériologie et la PCR en terme de détection des principales bactéries recherchées (H. somni, M. haemolytica et P. multocida). Tous les prélèvements négatifs en PCR pour ces agents, étaient également négatifs par culture. Les quelques discordances observées peuvent s’expliquer par la capacité de la PCR à détecter le génome de bactéries même non viables, et en parallèle les biais de compétition possible entre germes initialement présents dans un prélèvement lors de la mise en culture.
Tandis que la PCR offre la possibilité de détecter trois virus respiratoires des bovins, la bactériologie offre la possibilité de mettre en évidence des germes différents de ceux recherchés dans le kit PCR utilisé en routine à Labocea. En effet, sur les 53 prélèvements analysés, Streptococcus suis, Gallibacterium anatis, Bibersteinia trehalosi, Truperella pyogenes ont pu être isolés. À noter que du VRSB a été détecté en PCR sur ces mêmes échantillons.
Sur un poumon de bovin allaitant de quatre ans présentant de la toux et une dyspnée, la PCR était négative pour les sept agents, mais Aspergillus fumigatus a été isolé en culture pure.
En conclusion, l’une et l’autre des méthodes sont dans la majorité des cas concordantes sur les Pasteurellaceae recherchées dans le cadre des pathologies respiratoires des bovins. Utilisées conjointement, elles permettent d’apporter une réponse plus exhaustive sur l’ensemble des germes responsables de pathologie respiratoire.
Apports fondamentaux de l’autopsie et de l’histologie
La description des lésions d’autopsie et d’histologie (figure 2) permet de dater, d’évaluer l’étendue des lésions, d’orienter vers une implication plutôt bactérienne ou virale ou mixte mais aussi dans certains cas de conclure presque de manière définitive à l’implication d’un pathogène (Mh, Mb, IBR, dictyocaules) (14,15).
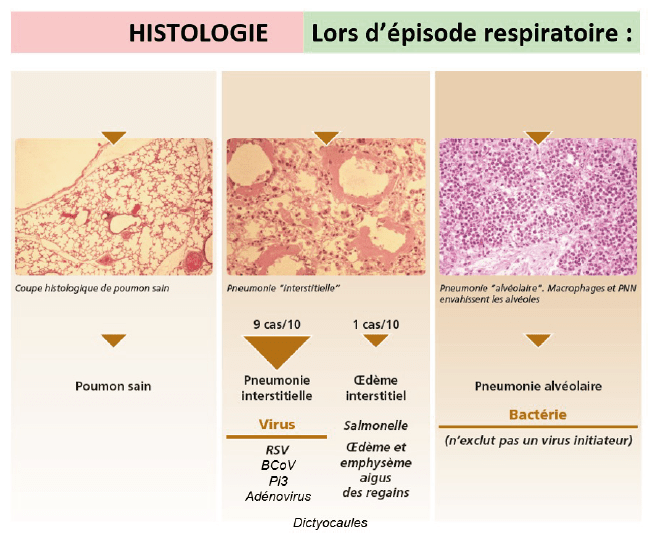
Les lésions de pneumonie alvéolaire signent l’implication des bactéries avec des lésions caractéristiques pour M. haemolytica (pleurésie fibrineuse, foyers de nécrose avec un liseré blanchâtre périphérique) et Mycoplasma bovis (abcès caséo-nécrotiques sans liseré). Ces deux pathogènes peuvent toutefois être isolés sans foyer de nécrose associé.
Lors d’infections virales, des lésions de bronchopneumonie interstielle sont observées. Contrairement à ce qui peut être pensé, il n’y a pas de lésions caractéristiques signant une infection par le VRSB. Des cellules syncytiales peuvent aussi être observées avec d’autres virus comme le Pi3, le BCoV, l’adénovirus…
L’emphysème pulmonaire et les lésions de pneumonies interstitielles ne sont pas non plus pathognomoniques d’une atteinte virale. Des lésions de pneumonie interstitielle sont aussi observées lors d’intoxications (emphysème des regains ou 3-methy-indol) ou d’infestation parasitaire (dictyocaules).
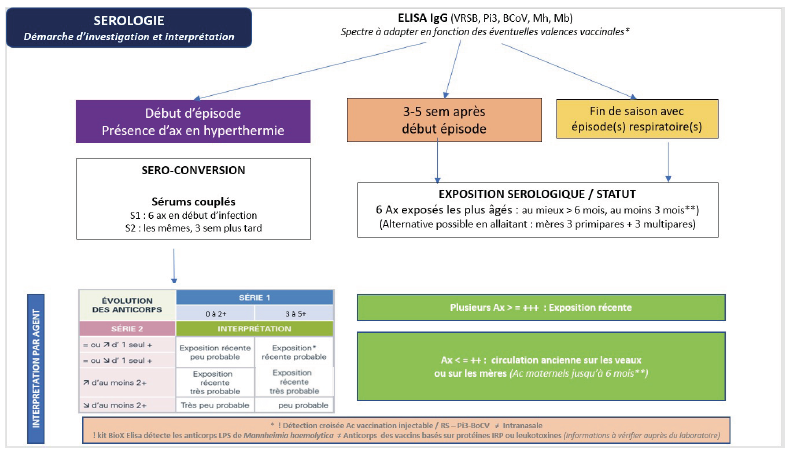
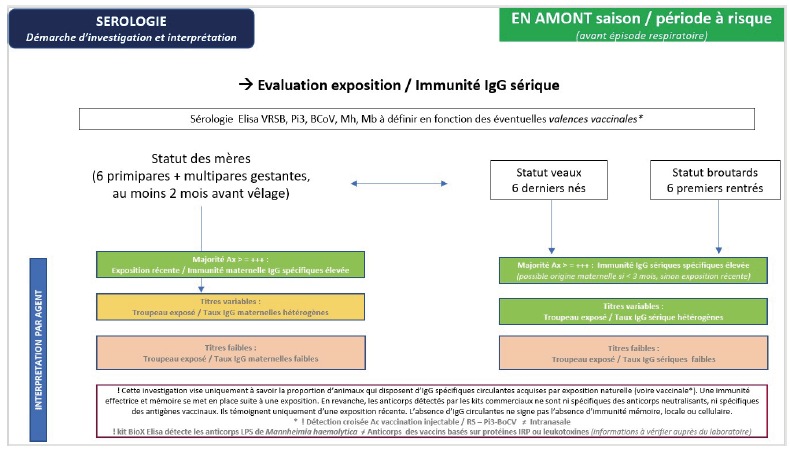
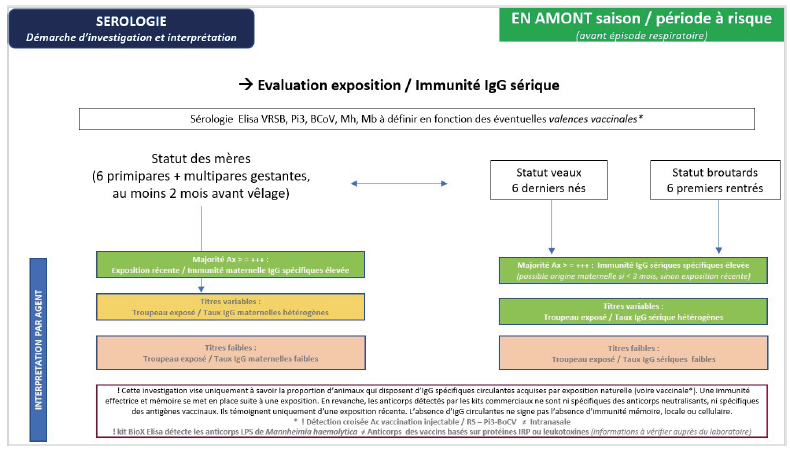
Intérêt de la sérologie pour investiguer l’exposition et le statut d’un troupeau
Le recours à la sérologie peut être une alternative intéressante pour rechercher une circulation d’un ou plusieurs agents primaires et/ou majeurs. L’interprétation doit nécessairement s’effectuer en fonction de plusieurs éléments, dont en premier lieu :
- Le moment de prélèvement par rapport au dernier épisode respiratoire
- Le statut immunitaire des animaux (immunité colostrale, exposition naturelle ou vaccinale antérieure)
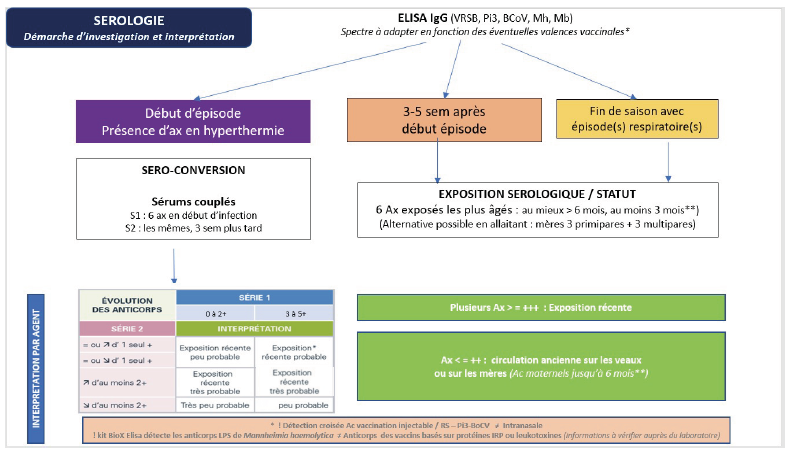
Pour interpréter l’implication d’un agent infectieux, il est préférable de mettre en évidence une séroconversion en réalisant une 1ère série de prélèvements en début d’infection (congénères en hyperthermie) et une 2ème série sur les mêmes animaux, trois semaines après (16).
Un des intérêts de la sérologie est aussi de pouvoir investiguer l’exposition des animaux vis-à-vis d’agents recherchés lors d’intervention tardive par rapport à l’épisode.
Dans ce cas, il est intéressant de prélever environ trois semaines après l’épisode 6 animaux exposés répondant aux critères suivants :
- les animaux exposés les plus âgés : au moins trois mois d’âge, mais de préférence de plus de six mois, pour limiter les titres élevés en anticorps maternels (alternative possible en allaitant : prélever les mères exposées à côté de leurs veaux),
- absence d’injection vaccinale sinon l’interprétation ne pourra porter que sur les valences concernées ou devra être prudente* si la dernière injection vaccinale remonte à moins de six mois.
Si ces conditions sont respectées, la mise en évidence de titres en anticorps spécifiques élevés (≥ +++ pour le kit BIOX K369 de Bio-X diagnostics®) sur la majorité des animaux est évocatrice d’une circulation récente des agents concernés.
La sérologie peut être également utilisée après une période ou saison à risque sur plusieurs animaux sentinelles > 6 mois d’âge et non vaccinés vis-à-vis des agents investigués* pour évaluer un statut du troupeau vis-à-vis de certains agents, sensibiliser les éleveurs et adapter le protocole de prévention.
Plusieurs propositions d’investigations sérologiques sont proposées (voir V).
*NB : Comment interpréter les résultats de l’ELISA pour des animaux vaccinés ?
Le risque de détection croisée des anticorps vaccinaux vis-à-vis des valences virales des vaccins injectable est élevé. Il est pour autant important de préciser qu’en dehors de validation spécifique, les tests ELISA commerciaux permettent rarement d’évaluer correctement une réponsevaccinale : ils ne sont pas spécifiques des souches ou des antigènes vaccinaux, ne ciblent pas particulièrement les anticorps neutralisants et n’évaluent pas la réponse cellulaire induite par des vaccins vivants atténués et certains vaccins inactivés adjuvés (17).
Par ailleurs, l’administration intranasale d’un virus vivant atténué non adjuvé peut induire une réponse mémoire systémique sans circulation d’anticorps au niveau sérique.
Aussi après une primo-infection naturelle ou une vaccination injectable, la demi-vie plasmatique des anticorps des agents respiratoires est relativement courte sans réexposition (18,19). Lors de nouvelle exposition naturelle ou vaccinale jusqu’à 1 an après, des anticorps peuvent être rapidement remis en circulation (17, 20).
Pour les agents bactériens, le risque de réponse croisée doit également être pris en compte et dépend des kits et des vaccins utilisés.
Par exemple, le principal kit multi-agents déployé en France (BioX K369) est basé sur un antigène LPS de Mannheimia haemolytica et ne détecte normalement pas d’anticorps induits par un vaccin dirigé contre les protéines IRP (17) ou la leucotoxine. Dans ce cas, sous réserve de vérification auprès du fabriquant de vaccin, l’interprétation sur cette valence reste donc possible malgré la vaccination.
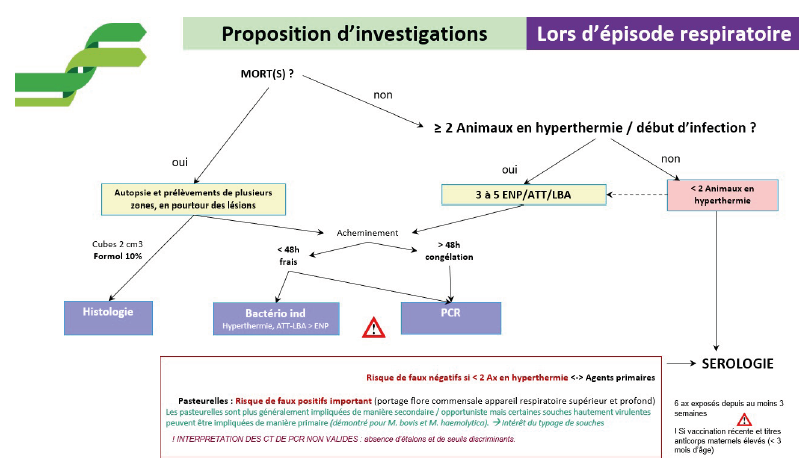
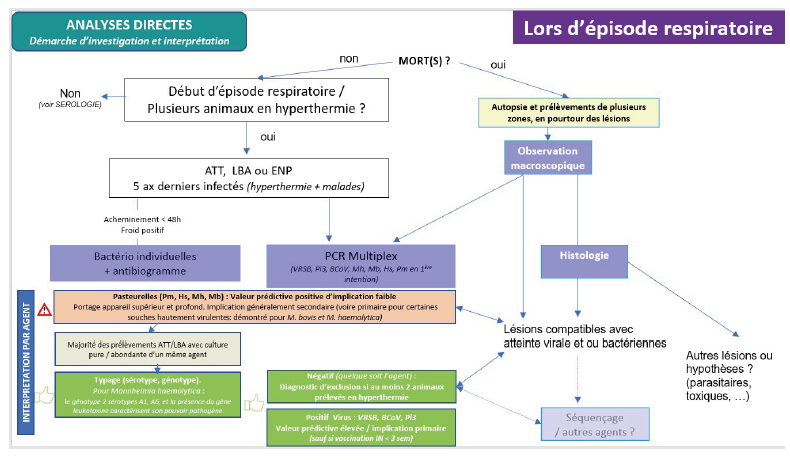
Conclusion : proposition de schémas d’investigations et d’interprétations
Il ne peut pas y avoir de « recette » unique mais obligatoirement une combinaison de différents facteurs à prendre en compte pour améliorer son diagnostic étiologique :
- Recueil clinique, épidémiologique et des facteurs de risques,
- Maîtrise des conditions de prélèvement : choix ciblé des animaux, constitution de mélanges, moment du prélèvement, choix de la matrice, etc…
- Si mortalité : autopsie et histologie de manière systématique
- Diagnostic direct si on est en cours d’épisode, indirect si l’intervention est plus tardive,
- Connaissance des intérêts et limites des méthodes,
La conclusion sur l’implication ou non d’un agent se fera en fonction de la combinaison des différents résultats obtenus et implique de prendre en compte les facteurs d’influence recueillis, les caractéristiques propres à chaque agent infectieux recherché (portage au niveau respiratoire, potentiel pathogène primaire/secondaire, de virulence et de diffusion) et l’immunité des animaux concernés (exposition antérieure, immunité maternelle, vaccination).
Les propositions de démarches simplifiées d’intervention en termes de diagnostic présentées ici ne se veulent pas exhaustives : en effet, le recensement de l’ensemble des cas auxquels le praticien peut être confronté n’est pas possible. Ces propositions pourraient constituer pour lui une « boîte à outils » que seule son expertise, le recueil des différentes données qu’il pourra effectuer et sa connaissance de l’élevage pourront valoriser. Plusieurs propositions d’investigations et d’interprétations associant analyses directes et/ou indirectes sont proposées en présence d’épisode respiratoire, en amont d’une période à risque (avant les 1ers cas) ou après une saison à risque (schémas 1 à 4).
Bibliographie
1. PARDON B., BUCZINSKI S. Bovine Respiratory Disease Diagnosis: What Progress Has Been Made in Infectious Diagnosis? Vet Clin North Am Food Anim Pract. 2020; 36(2):425-444.
2. PARDON B., CALLENS J., MARIS J. ALLAIS L., VAN PRAET W., DEPREZ P., RIBBENS S. Pathogen-specific risk factors in acute outbreaks of respiratory disease in calves. J Dairy Sci. 2020; 103(3):2556-2566.
3. VAN DRIESSCHE L., VALGAEREN B., R., GILLE L., BOYEN F., DUCATELLE R., HAESEBROUCK F., PARDON B. A deep nasopharyngeal swab versus nonendoscopic bronchoalveolar lavage for isolation of bacterial pathogens from preweaned calves with respiratory disease. Journal of veterinary internal medicine. 2017; 31(3):946-953.
4. THOMAS A.C., BAILEY M., LEE M. MEAD A., MORALES-AZA B., REYNOLDS R., VIPOND B., FINN A., EISLER M.C. Insights into Pasteurellaceae carriage dynamics in the nasal passages of healthy beef calves. Sci Rep. 2019; 9(1):11943.
5. ANGEN O., THOMSEN J., LARSEN L.E. Respiratory disease in calves: microbiological investigations on trans-tracheally aspirated bronchoalveolar fluid and acute phase protein response. Vet Microbiol. 2009; 137(1-2):165-171
6. TIMSIT E., HALLEWELL J., BOOKER C., TISON N., AMAT S., ALEXANDER T.W. Prevalence and antimicrobial susceptibility of Mannheimia haemolytica, Pasteurella multocida, and Histophilus somni isolated from the lower respiratory tract of healthy feedlot cattle and those diagnosed with bovine respiratory disease. Vet Microbiol. 2017; 208:118-125.
7. LOWIE T., CALLENS J., MARIS J., RIBBENS S., PARDON B. Decision tree analysis for pathogen identification based on circumstantial factors in outbreaks of bovine respiratory disease in calves. Prev Vet Med. 2021; 196:105469.
8. TIMSIT E., CHRISTENSEN H., BAREILLE N., SEEGERS H., BISGAARD M., ASSIE S. Transmission dynamics of Mannheimia haemolytica in newly-received beef bulls at fattening operations. Vet Microbiol. 2013; 161(3-4):295-304.
9. TIMSIT E., ARCANGIOLI M.A., BAREILLE N., SEEGERS H., ASSIE S. Transmission dynamics of Mycoplasma bovis in newly received beef bulls at fattening operations. J Vet Diagn Invest. 2012; 24(6):1172-1176.
10. MEYER G., PELLETIER C., HERMAN N., DUCATEZ M., CASSARD H., SALEM E. Vers une identification de nouveaux virus respiratoires bovins. Nouveau Praticien Vétérinaire. 2015 ; 31(8):8-14.
11. JOZAN T., MELINE B. Vacciner les mères et les jeunes veaux dès leur plus jeune âge. L’exemple de la prévention des maladies respiratoires. Proceeding JNGTV. 2018 ; 655-660.
12. TIMSIT E., LE DREAN E., MAINGOURD C., BELLOC C., GUATTEO R., BAREILLE N., SEEGERS H., DOUART A., SELLAL E., ASSIE S. Detection by real-time RT-PCR of a bovine respiratory syncytial virus vaccine in calves vaccinated intranasally. Vet Rec. 2009; 165(8):230-233.
13. KLOMPMAKER A.F., BRYDENSHOLT M., MICHELSEN A.M., et al. Estimating Clinically Relevant Cut-Off Values for a High-Throughput Quantitative Real-Time PCR Detecting Bacterial Respiratory Pathogens in Cattle. Front Vet Sci. 2021; 8:674771.
14. Guide pratique de diagnostic. 2ème édition juillet 2015 réalisé par INP Toulouse et MSD Santé Animale.
15. PANCIERA R.J., CONFER A.W. Pathogenesis and pathology of bovine pneumonia. Vet Clin North Am Food Anim Pract. 2010;26(2):191-214.
16. FULTON R.W., CONFER A.W. Laboratory test descriptions for bovine respiratory disease diagnosis and their strengths and weaknesses: gold standards for diagnosis, do they exist ?. Can Vet J. 2012; 53(7):754-761.
17. BERGE A. C., JOZAN T., LEVESQUE C., VERTENTEN G. A Field Study Evaluating Humoral Immunity in Calves Vaccinated with Multivalent Bovine Respiratory Pathogen Vaccines. Folia Veterinaria. 2021; 65(4):20-30.
18. KIMMAN T. G., WESTENRINK F., SCHREUDER B. E., STRAVER P.J. Local and systemic antibody response to bovine respiratory syncytial virus infection and reinfection in calves with and without maternal antibodies. J. Clin. Microbiol. 1987; 25:1097-1106.
19. KIMMAN T. G., WESTENBRINK F., STRAVER P. J. Priming for local and systemic antibody memory responses to bovine respiratory syncytial virus : effect of amount of virus, virus replication, route of administration and maternal antibodies. Vet. Immunol. Immunopathol. 1989; 22:145—160.
20. MAKOSCHEY B., BERGE A. C. Review on bovine respiratory syncytial virus and bovine parainfluenza—usual suspects in bovine respiratory disease—A narrative review. BMC Vet. Res. 2021; 17:261—279.
21. LOY J., CLAWSON M. Rapid typing of Mannheimia haemolytica major genotypes 1 and 2 using MALDI-TOF mass spectrometry. Journal of Microbiological Methods.2017;136:30-33
22. KLIMA C., ZAHEER R., BRIGGS R., McALLISTER T. A multiplex PCR assay for molecular capsular serotyping of Mannheimia haemolytica serotypes 1, 2, and 6. Journal of Microbiological Methods.2017; 139 :155-160.
23. COZENS D., SUTHERLAND E., LAUDER M., TAYLOR G. BERRY C., DAVIES R. Pathogenic Mannheimia haemolytica invades differentiated bovine airway epithelial cells. Infection and Immunity. 2019; 87(6): e00078-19.
24. NAKAY I., NAKAZAWA M., HASHIMOT Y., IKEUCHI T., NOMA S., TORIKAI Y., EZAKI T. Isolation and characterization of Pasteurella trehalosi from cattle. Journal of the Japan Veterinary Medical Association. 1995; 48:750–754.
25. COLLINS RL. Bibersteinia trehalosi in cattle – another component of the bovine respiratory disease complex? Cattle Practice.2011;19:9–12.
26. BROWN S., BYCROFT K., ADAM K., COLLETT M. Acute fibrinous pleuropneumonia and septicaemia caused by Bibersteinia trehalosi in neonatal calves in New Zealand. New Zealand Veterinary Journal. 2021; 69(1):51-57.
27. CORTESE V., BRAUN D., CROUCH D., TOWNSEND C., ZUKOWSKI B. Case report – peracute to acute fatal pneumonia in cattle caused by Bibersteinia trehalosi. Bovine Practitioner. 2012;46,138–42.
28. BOWERSOCK T., SOBECKI B., TERRILL S., MARTINON N., MEINERT T., LEYH R. Efficacy of a multivalent modified live virus vaccine containing a Mannheimia haemolytica toxoid in calves challenge exposed with Bibersteinia trehalosi. American Journal of Veterinary Research. 2014; 75:770-776.
29. VAN DRIESSCHE L., VANNESTE K., BOGAERTS B., DE KEEESMAECKER S., Nancy H. ROOSENS N., HAESEBROUCK F., DE CREMER L., DEPREZ P., PARDON B., BOYEN F. Isolation of Drug-Resistant Gallibacterium anatis from Calves with Unresponsive Bronchopneumonia, Belgium. Emerging Infectious Diseases. 2020; 26:721-730.
GP-FR-NON-220400025
Exemples de cas et propositions de schémas d’interprétation.
Auteurs :
Dr Camille Levesque, chef de service Virologie et Sérologie au LABOCEA 35, camille.levesque@labocea.fr
Dr Guillaume Lequeux, chef de service Bactériologie et parasitologie au LABOCEA 35, guillaume.lequeux@labocea.fr
Dr Thibault Jozan, vétérinaire responsable technique MSD Santé Animale.
Introduction
De nouveaux outils de diagnostic ont été développés et sont proposés par les laboratoires : identifications bactériennes par spectrométrie de masse MALDI-TOF, PCR, séquençage, ou de façon indirecte par méthode ELISA. Cependant, l’interprétation des résultats d’analyse obtenus peut être problématique car la détection des agents peut dépendre du type de prélèvement (Ecouvillon Nasal Profond – ENP, Aspiration TransTrachéale – ATT, Lavage Broncho-Alvéolaire – LBA, Poumon), du nombre d’animaux prélevés, du délai entre l’apparition des premiers symptômes et le prélèvement, de l’immunité des animaux et des limites propres à chaque méthode d’analyse.
De plus, l’implication de certains pathogènes dans les pathologies respiratoires fait toujours l’objet de controverses (1,2). En effet, en dehors des agents primaires reconnus, la détection d’agents dits « secondaires » pose question. Or, ces agents opportunistes peuvent être de simples contaminants, appartenir à la flore commensale de l’appareil respiratoire des animaux sains (3,4,5,6) ou encore se multiplier à la faveur de conditions favorisantes infectieuses ou non, d’une baisse d’immunité et participer à la dégradation de la santé de l’animal.
En complément de l’identification d’agents avec une bonne sensibilité et spécificité, les outils diagnostiques doivent permettre une différenciation entre une contamination (lors de la réalisation du prélèvement par exemple), portage (présence sans inflammation, ou modification de l’état de santé de l’animal) et infection de l’appareil respiratoire avec répercussion subclinique ou clinique. Est-ce que l’association de différentes analyses peut pallier les limites rencontrées à l’heure actuelle dans l’interprétation de certains résultats ?
Les principaux intérêts et limites des analyses de laboratoire les plus courantes seront présentés ainsi que des exemples d’associations d’analyses réalisées à Labocea et d’interprétation des résultats. Enfin, des schémas d’aide pour le choix des analyses à réaliser en fonction de la situation épidémiologique et de l’objectif recherché sont proposés.
Intérêts et limites de la PCR
Lors d’épisode respiratoire, le recours aux analyses PCR est fréquent.
A Labocea, entre 2017 et 2022, près de 1400 analyses de PCR temps-réel multi-agents avec le kit VetMAX™ Ruminant Respiratory Screening Kit (Thermofisher scientific) permettant la mise en évidence du génome de quatre bactéries (Mannheimia haemolytica (Mh), Pasteurella multocida (Pm), Histophilus somni (Hs) et Mycoplasma bovis (Mb)) et trois virus (VRSB, Pi3 et Coronavirus bovin (BCoV)) simultanément, ont été réalisées. Les PCR développées et validées sont en général considérées comme très sensibles (seuil de détection bas et capacité à détecter des bactéries ou virus viables ou non). Cela se traduit par la détection dans une grande majorité des cas d’au moins un agent par analyse réalisée. Ainsi, à Labocea, la proportion d’analyses avec au moins un agent détecté est de 92 % entre janvier 2017 et janvier 2022 avec la PCR multi-agents.
La PCR bénéficie d’une très bonne sensibilité, y compris sur mélange de cinq prélèvements de nature identique. En augmentant le nombre d’animaux prélevés, le risque de faux négatifs par rapport aux agents recherchés est limité (2).
D’après les résultats obtenus à Labocea, à l’exclusion d’Histophilus somni, la fréquence de détection des autres agents recherchés est significativement plus importante (p<0.005) par les analyses de mélanges de deux à cinq écouvillons (tableau 1).
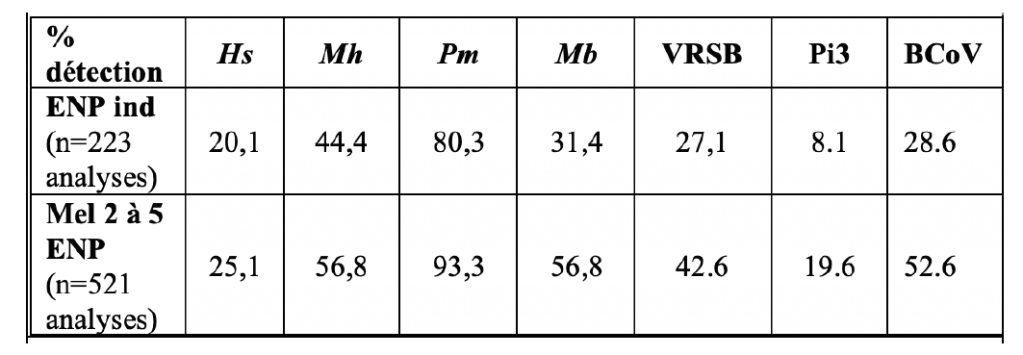
PCR temps-réel multi-agents avec le kit VetMAX™ Ruminant Respiratory Screening Kit (Thermofisher scientific) permettant la mise en évidence du génome de 4 bactéries (Mannheimia haemolytica (Mh), Pasteurella multocida (Pm), Histophilus somni (Hs) et Mycoplasma bovis (Mb)) et 3 virus (VRSB, Pi3 et Coronavirus bovin (BCoV).
La fréquence de détection des différents agents pathogènes peut différer selon la matrice prélevée (1,2,3,7) avec des observations parfois contradictoires. Lowie T. et al. (7) indiquent une meilleure détection du BCoV et du VRSB sur lavage broncho-alvéolaire LBA que sur ENP. Dans une étude de Meyer et al. (10) dans laquelle des ENP et LBA ont été réalisés en parallèle sur les mêmes veaux, le VRSB est détecté 4x plus sur LBA alors que le BCoV est détecté 1,5 fois plus sur ENP.
D’après les résultats obtenus au laboratoire depuis 2017, la fréquence de détection du Coronavirus bovin est significativement différente (p<0.001) sur les prélèvements individuels d’ENP (28.6 %) par rapport aux poumons (10.7 %). De même, Mannheimia haemolytica et Pasteurella multocida ont des fréquences de détection apparentes significativement différentes (p<0.001) entre ENP individuel (Mh : 44 %, Pm 80 %) et poumons (Mh 61 % et Pm 57 %).
Attention, dans les données de Labocea présentées ci-dessus (comparaison selon la matrice et le nombre d’échantillons analysés dans le mélange), s’agissant de prélèvements dans des élevages différents, les différences peuvent être potentiellement le reflet de contextes épidémiologiques cliniques différents, ayant conduit à des choix de prélèvements différents.
Par ailleurs, comme pour toute analyse de laboratoire, le délai entre la date d’apparition des premiers symptômes et le prélèvement est à prendre en compte dans l’interprétation des résultats d’analyses.
Lorsque le vétérinaire intervient, il n’est pas rare que le début de l’épisode remonte déjà à plusieurs jours, diminuant ainsi fortement la valeur prédictive de l’implication ou non des agents recherchés par analyses directes (11) :
- un risque de faux négatifs vis-à-vis d’agents primaires ou initiateurs (2,7).
Les virus et certaines souches bactériennes à pouvoir pathogène ou de diffusion élevé, comme démontré pour M. haemolytica et M. bovis (8,9) ont un rôle primaire important lors d’épisode respiratoire d’allure épidémique. Les infections virales sont sous-détectées lorsque les prélèvements ne peuvent pas être réalisées en début d’infection (2,7).
Lors de mortalité, il n’est pas rare de retrouver uniquement des pasteurelles sur des PCR de poumon alors que l’intervention d’un virus est démontrée par la réalisation d’analyses complémentaires comme l’histologie et/ou des sérologies sur les congénères (voir III et IV).
Il paraît donc important de prélever plusieurs animaux en début d’infection afin de limiter les risques de ne pas détecter les agents primaires ou initiateurs (2,7,8,9).
La connaissance de traitements ou de vaccination antérieures est également à prendre en compte dans l’interprétation des résultats. En effet, il existe également :
- un risque de faux négatifs vis-à-vis des bactéries lors de traitement antibiotique, même si ce risque est beaucoup plus limité qu’en culture bactériologique,
- un risque de faux positifs pour VRS et Pi3 lors de vaccination avec des agents vivants atténués en intranasal. Des faux positifs vis-à-vis de ces agents sont possibles par les méthodes PCR jusqu’à 1 mois après la vaccination pour VRSB (12).
NB : La détection des agents couverts par un vaccin ne suffit pas en elle-même à caractériser une immunodéficience ou un échec vaccinal.
Les différents vaccins respiratoires du marché peuvent obtenir des indications de réduction de l’excrétion, de la clinique, voire des lésions propres à chaque vaccin et chaque valence. La vaccination vise donc à réduire les conséquences d’une infection vis-à-vis des principaux agents représentant le plus grand risque. Il n’est pas anormal de retrouver par PCR des agents ciblés par le protocole vaccinal chez les animaux correctement immunisés, en bonne santé.
Enfin, l’interprétation vis-à-vis des pasteurelles reste délicate :
la détection par PCR de Hs, Mh, Pm ou même Mb, du fait de leur portage au niveau de la flore de l’appareil respiratoire supérieur et profond pose la question de l’interprétation des résultats positifs pour ces agents (encadré 1).
Encadré 1 : L’appareil respiratoire profond n’est pas stérile !
Quid de l’identification de pasteurelles sur poumon, aspiration transtrachéale (ATT), lavage broncho-alvéolaire (LBA) et écouvillon nasal profond (ENP) en pratique ?
Le temps où l’on pensait que le seul diagnostic de pasteurelles au niveau respiratoire profond permettait de signer leur implication au moins secondaire est révolu.
De nombreuses publications (2,4,5,6,13,14) rapportent la détection fréquente de pasteurelles par PCR ou bactériologie sur des prélèvements ATT ou LBA en l’absence d’épisode respiratoire et/ou sur des animaux « sains » au niveau clinique, échographique et/ou des marqueurs de l’inflammation.
Dans l’étude de Angen O. et al. (5), des aspirations transtrachéales ont été réalisées sur des veaux d’élevages laitiers visités en été en l’absence d’épisode respiratoire, puis en hiver lors d’épisode respiratoire. Des pasteurelles ont été identifiées chez 68 % des veaux prélevés par ATT en l’absence d’épisode respiratoire ; 59 % par culture et 41 % par PCR. Un élevage sur deux avait plus de 30 % d’ATT positifs vis-à-vis de Histophilus somni, 4/6 avec plus de 50 % pour Pasteurella multocida. Pour Mannheimia haemolytica, cinq élevages sur six avaient moins de 20 % d’ATT positifs (tableau 1). En pratique, la réalisation de PCR par pool de trois à cinq prélèvements augmente cette probabilité de détection déjà élevée en l’absence de maladie.
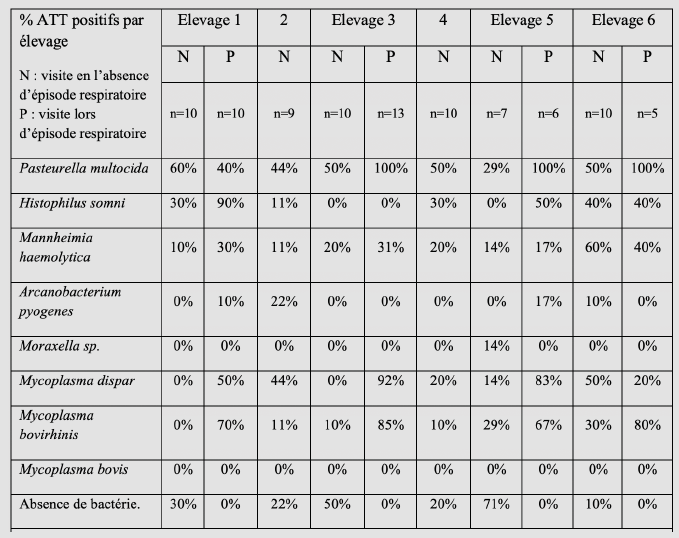
Est-ce qu’une analyse quantitative permettrait de faire la distinction entre la colonisation sans conséquence et la surinfection avec dégradation de l’état de santé ?
Une étude de Klompmaker et al. (13) en particulier a été menée dans ce sens. Pour H. somni, un CT ≤ 17,4 était associé à 86 % des veaux malades mais un CT > 17,4 était aussi associé à 40 % de veaux malades. L’établissement de seuil ne semble pas suffisamment discriminant dans la mesure où une proportion relativement importante d’animaux classés comme sains pouvait avoir un CT inférieur au seuil choisis, en particulier pour P. multocida et M. bovis. Aucun seuil n’a pu être établi pour T. pyogenes et Mannheimia (figure 1). Les auteurs précisent qu’il conviendrait de distinguer les différents sérotypes de M. haemolytica afin de prendre en compte leurs différences de pouvoir pathogène (encadré 2).
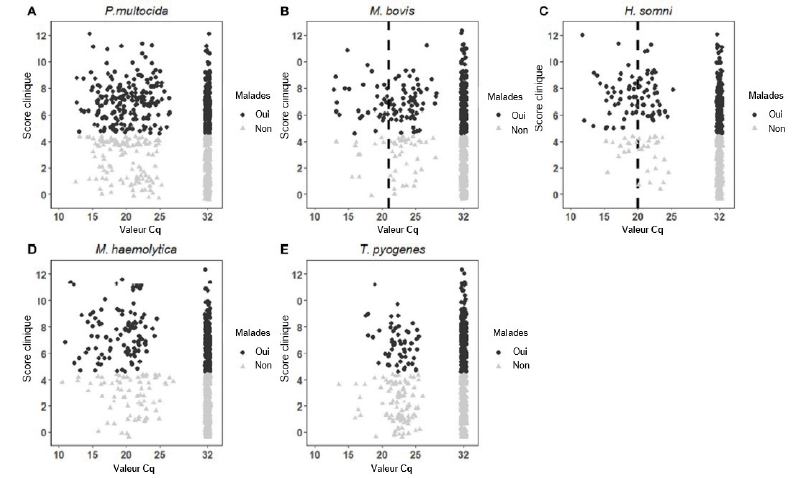
L’établissement de seuil ne semble pas une piste suffisamment aboutie à ce jour, ce qui est corroboré aussi par l’étude de Thomas et al. (4). Cette étude présente les valeurs obtenues en PCR quantitatives de portages asymptomatiques des Pasteurellaceae sur des écouvillons de veaux allaitants sevrés, cliniquement sains sur une durée de 75 jours. Ces veaux avaient été allotés 2 mois avant le début des prélèvements et n’ont pas été déplacés. 95 % des veaux étaient plusieurs fois positifs pour P. multocida, 75 % pour H. somni et 13 % pour M. haemolytica. La distribution des densités bactériennes mesurées par qPCR était très étendue (figure 2).
À ce jour, aucune donnée et méthode ne permettent d’établir de lien fiable et valorisable en pratique entre le CT et l’imputabilité de l’agent détecté.
Une des dernières limites de la PCR est qu’on ne peut détecter que ce que l’on recherche a priori et en fonction des kits disponibles. D’autres agents tels que le BCoV ou l’influenza D ne font pas toujours partie des kits utilisés en routine en première intention alors que leur implication dans le complexe respiratoire bovin parait fréquente et est de plus en plus reconnue (2,10).
Au vu des limites de la PCR, il apparaît alors pertinent d’associer la PCR à d’autres méthodes d’analyses (séquençage, bactériologie, MALDI-TOF, sérologie). Des exemples d’associations de la PCR avec d’autres méthodes sont présentés ci-après (encadrés 2 et 3).
Intérêts et limites de la bactériologie
L’interprétation de la bactériologie se fait en tenant compte de différents facteurs, comme pour la PCR (conditions de prélèvements et échantillonnage, matrice, traitements antibiotiques éventuels).
Il est important de tenir compte :
- des commémoratifs cliniques et épidémiologiques et d’investigation des facteurs de risques qui permettent de fortement orienter vers une étiologie plutôt qu’une autre (7),
- du potentiel de chaque agent d’agir comme pathogène primaire ou secondaire par rapport à son portage au niveau de la sphère respiratoire supérieure et profonde (1,2).
La prise en compte des éléments de pureté et d’abondance des colonies est capitale à l’interprétation pour toutes les bactéries.
On pourrait penser que l’identification sur plusieurs prélèvements ATT/LBA d’animaux en début d’infection, d’une même pasteurelle en culture pure et abondante présage de son implication au moins secondaire. Il convient toutefois de rester prudent. Van Leenen et al. (3) n’ont pas mis en évidence de différence de taux d’isolement bactérien et des résultats quantitatifs entre les cultures bactériologiques réalisées sur les LBA de veaux sains et ceux diagnostiqués en infection respiratoire subclinique ou clinique.
En complément de la culture bactériologique, la recherche de facteur de virulence par PCR ou MALDI-TOF ou séquençage permet de différencier des souches selon leur potentiel pathogène ou de résistance. Ces investigations complémentaires se déploient en laboratoire d’analyse et permettent d’apporter de nouvelles données intéressantes pour interpréter les résultats. Pour Mannheimia haemolytica, il est maintenant possible de déterminer le génotype et sérotype d’une souche ainsi que la présence ou non du gène de la leucotoxine. Ces éléments informatifs sont intéressants pour interpréter les résultats (encadré 2). Dans une étude réalisée chez des jeunes bovins à l’engraissement en Pays de la Loire, 63 % des souches isolées correspondaient à du sérotype A6. Les sérotypes A1 et A2 ne représentaient que 30 % et 7 % des cas respectivement (8).
Encadré 2 : Quelles informations apporte la détermination du génotype et du sérotype des souches de M. haemolytica isolées en pathologie respiratoire ?
Les outils diagnostiques permettant une caractérisation plus fine des souches de M. haemolytica impliquées en pathologie respiratoire bovine sont à présent relativement facilement accessibles en routine :
- la détermination du génotype est permise lors de l’identification des souches par spectrométrie de masse MALDI-TOF, méthode d’identification qui se généralise et devient courante en diagnostic vétérinaire (20),
- la détermination du sérotype peut être faite par PCR temps-réel, méthode qui s’est substituée au sérotypage par agglutination (21). Les PCR temps-réel actuellement disponibles permettent également de détecter sur les souches isolées la présence du gène codant pour la leucotoxine (LTx), principal facteur de virulence. La détection de ce gène sur une souche isolée en contexte clinique et/ou lésionnel permet de lui attribuer avec une forte probabilité l’origine des symptômes et/ou lésions observées.
Une étude américaine récente concernant Mannheimia haemolytica a permis de montrer que seules les souches appartenant au génotype 2 peuvent être considérées comme pathogènes (car systématiquement retrouvées dans les poumons d’animaux malades, au contraire du génotype 1 qui correspond aux souches commensales retrouvées exclusivement dans le nasopharynx d’animaux sains). Par ailleurs, les supports de résistance voire de multirésistance aux antibiotiques (intégrons) sont exclusivement retrouvés chez les souches de génotype 2 (20).
Des auteurs ont réussi à déterminer qu’il existe des pics spécifiques à chacun de ces deux génotypes en spectrométrie de masse MALDI-TOF, permettant ainsi de les différencier de façon rapide, sans nécessité d’effectuer un génotypage complet (20). Ces travaux ouvrent la possibilité pour le praticien de connaître dès le lendemain de la réception d’un échantillon au laboratoire le génotype d’une souche de M. haemolytica qui serait isolée et donc son pouvoir pathogène mais également son profil de résistance probables.
Dans la séquence pathogénique des infections à M. haemolytica, il a été démontré que les bovins sains sont le plus souvent colonisés par des souches commensales de sérotype A2, mais que les infections respiratoires sont le plus fréquemment causées par des souches pathogènes appartenant au sérotype A1 (22). Les raisons de cette transition de populations bactériennes majoritairement de sérotype A2 vers une « explosion » de populations bactériennes de sérotype A1 dans le tractus respiratoire ne sont pas complètement connues, mais il semblerait que seules les souches de sérotype A1 possèdent des facteurs de virulence permettant l’invasion puis leur prolifération à l’intérieur des cellules de l’épithélium respiratoire (22).
Aussi l’avènement de l’identification bactérienne par MALDI-TOF a permis de mettre en évidence de façon plus simple, fiable et rapide, d’autres pasteurelles potentiellement impliquées en pathologies bovines.
Deux espèces appartenant à la famille des Pasteurellaceae ont notamment fait l’objet de descriptions récentes (< 10 ans) en tant qu’agents impliqués en pathologie bovine : Bibersteinia trehalosi et Gallibacterium anatis.
Bibersteinia trehalosi est une bactérie principalement décrite comme agent responsable de septicémies aigües (« pasteurellose systémique ») chez les agneaux de plus de 6 mois. Cette espèce avait déjà été décrite comme commensal possible du nasopharynx, des conjonctives oculaires et des amygdales chez les bovins (23).
Chez les bovins, différentes présentations cliniques lors d’infections à Bibersteinia trehalosi sont décrites particulièrement depuis une dizaine d’années : hépatite nécrosante et sérosite fibrineuse chez des vaches adultes, bronchopneumonies fibrino-purulentes voire nécrosantes chez des bovins de plus de mois (24). Des cas de pleuropneumonies associées à des septicémies ont également récemment été décrits chez des veaux laitiers nouveau-nés (25).
Bibersteinia trehalosi est à présent reconnu en Amérique du Nord comme agent bactérien émergent, pathogène primaire impliqué dans le complexe respiratoire bovin chez les bovins en engraissement mais aussi chez de jeunes veaux (26). Cet agent est de plus en plus isolé dans ces contextes cliniques depuis 2007 environ.
Il est possible, selon les auteurs de ces différentes études, que l’implication de Bibersteinia trehalosi en pathologie bovine ait été sous-estimée pendant des années du fait des difficultés existantes à la différencier de M. haemolytica lors de son identification bactériologique, alors que les tableaux lésionnels liés à ces deux bactéries ne sont pas différenciables. L’avènement de l’identification bactérienne par spectrométrie de masse MALDI-TOF, permettant une identification facile et fiable de cette espèce bactérienne pourrait être une explication de l’augmentation de son incidence dans l’espèce bovine.
Il est intéressant de noter que M. haemolytica et Bibersteinia trehalosi partagent des facteurs de virulence communs (lipopolysaccharides et leucotoxine) et que l’utilisation de vaccins dirigés contre M. haemolytica a démontré une protection croisée contre les infections à Bibersteinia trehalosi chez les bovins (27).
Enfin, les études de séquençage tendent à démontrer que, dans les troupeaux mixtes ovins-bovins, la transmission de cette bactérie se ferait plutôt dans le sens bovins vers ovins que l’inverse (25).
En France, peu de données existent, mais il est constaté un à deux isolements par an depuis 2017 environ à LABOCEA Fougères alors que de tels isolements n’avaient pas été constatés précédemment. Il est néanmoins possible, là encore, que ce constat puisse en partie être lié à l’utilisation en routine du MALDI-TOF pour les identifications bactériennes depuis quelques années.
Gallibacterium anatis a fait l’objet d’une description beaucoup plus récente. Il s’agit d’une bactérie émergente chez les volailles (espèce poule en particulier), pathogène opportuniste responsable principalement de septicémies (salpingites, péritonites, épididymites et lésions respiratoires) dans cette espèce.
Gallibacterium anatis avait précédemment été isolée à partir de fèces chez des bovins sains, mais une récente étude en Belgique suggère de considérer cette bactérie comme pathogène respiratoire émergent, secondaire et opportuniste chez les bovins (28). Dans cette publication, aucun lien n’avait pu être fait avec une contamination éventuelle à partir d’élevages aviaires. Il semble que, comme précédemment décrit pour Bibersteinia trehalosi, l’incidence de Gallibacterium anatis chez les bovins ait augmenté ces dernières années du fait d’une utilisation plus large du MALDI-TOF, outil qui aurait seul ou en grande parti augmenté le taux d’identification de cette bactérie.
La préoccupation majeure, selon ces auteurs (28) concernant Gallibacterium anatis réside dans le constat (partagé par les observations faites à LABOCEA Fougères) de fréquentes multi-résistances chez les souches isolées de pathologies respiratoires bovines, dont des résistances fréquentes aux fluoroquinolones.
Enfin, la bactériologie reste incontournable pour réaliser des antibiogrammes et suivre régulièrement l’évolution des sensibilités des antibiotiques du protocole thérapeutique vis-à-vis des pasteurelles.
Encadré 3 : Association des analyses PCR 7 agents respiratoires et de la bactériologie classique.
Analyses des données comparées des forfaits respiratoires réalisés à Labocea.
53 analyses ont été réalisées en parallèle en bacteriologie et en PCR entre décembre 2020 et février 2022 à partir du même prélèvement de départ. La bactériologie consistait à réaliser une culture bactérienne sur milieux sélectifs et non sélectifs puis à identifier les colonies d’intérêt par MALDI-TOF (à l’exclusion de Mycoplasma bovis).
Pour la PCR, le kit VetMAX™ Ruminant Respiratory Screening Kit (Thermofisher scientific) est utilisé et permet la recherche des 7 agents simultanément : Histophilus somni (H. somni), Mannheimia haemolytica (M. haemolytica), Pasteurella multocida (P. multocida), Mycoplasma bovis (M. bovis), Coronavirus bovin (BCoV), Parainfluenza 3 (Pi3) et le virus respiratoire syncitial bovin (VRSB).
Les prélèvements reçus sont principalement des écouvillons nasaux pharyngés (31 dossiers), puis des poumons (n= 15), ATT (4) et LBA (3). Les demandes d’analyse de mélanges sont majoritaires pour les ENP reçus. La moitié des demandes au moins concernent des bovins âgés entre un et six mois.
Pour 50 analyses sur 53 réalisées au moins un agent microbien potentiellement pathogène a été détecté. De plus, 67 % des prélèvements analysés ont obtenus des résultats concordants entre la bactériologie et la PCR en terme de détection des principales bactéries recherchées (H. somni, M. haemolytica et P. multocida). Tous les prélèvements négatifs en PCR pour ces agents, étaient également négatifs par culture. Les quelques discordances observées peuvent s’expliquer par la capacité de la PCR à détecter le génome de bactéries même non viables, et en parallèle les biais de compétition possible entre germes initialement présents dans un prélèvement lors de la mise en culture.
Tandis que la PCR offre la possibilité de détecter trois virus respiratoires des bovins, la bactériologie offre la possibilité de mettre en évidence des germes différents de ceux recherchés dans le kit PCR utilisé en routine à Labocea. En effet, sur les 53 prélèvements analysés, Streptococcus suis, Gallibacterium anatis, Bibersteinia trehalosi, Truperella pyogenes ont pu être isolés. À noter que du VRSB a été détecté en PCR sur ces mêmes échantillons.
Sur un poumon de bovin allaitant de quatre ans présentant de la toux et une dyspnée, la PCR était négative pour les sept agents, mais Aspergillus fumigatus a été isolé en culture pure.
En conclusion, l’une et l’autre des méthodes sont dans la majorité des cas concordantes sur les Pasteurellaceae recherchées dans le cadre des pathologies respiratoires des bovins. Utilisées conjointement, elles permettent d’apporter une réponse plus exhaustive sur l’ensemble des germes responsables de pathologie respiratoire.
Apports fondamentaux de l’autopsie et de l’histologie
La description des lésions d’autopsie et d’histologie (figure 2) permet de dater, d’évaluer l’étendue des lésions, d’orienter vers une implication plutôt bactérienne ou virale ou mixte mais aussi dans certains cas de conclure presque de manière définitive à l’implication d’un pathogène (Mh, Mb, IBR, dictyocaules) (14,15).

Les lésions de pneumonie alvéolaire signent l’implication des bactéries avec des lésions caractéristiques pour M. haemolytica (pleurésie fibrineuse, foyers de nécrose avec un liseré blanchâtre périphérique) et Mycoplasma bovis (abcès caséo-nécrotiques sans liseré). Ces deux pathogènes peuvent toutefois être isolés sans foyer de nécrose associé.
Lors d’infections virales, des lésions de bronchopneumonie interstielle sont observées. Contrairement à ce qui peut être pensé, il n’y a pas de lésions caractéristiques signant une infection par le VRSB. Des cellules syncytiales peuvent aussi être observées avec d’autres virus comme le Pi3, le BCoV, l’adénovirus…
L’emphysème pulmonaire et les lésions de pneumonies interstitielles ne sont pas non plus pathognomoniques d’une atteinte virale. Des lésions de pneumonie interstitielle sont aussi observées lors d’intoxications (emphysème des regains ou 3-methy-indol) ou d’infestation parasitaire (dictyocaules).
Intérêt de la sérologie pour investiguer l’exposition et le statut d’un troupeau
Le recours à la sérologie peut être une alternative intéressante pour rechercher une circulation d’un ou plusieurs agents primaires et/ou majeurs. L’interprétation doit nécessairement s’effectuer en fonction de plusieurs éléments, dont en premier lieu :
- Le moment de prélèvement par rapport au dernier épisode respiratoire
- Le statut immunitaire des animaux (immunité colostrale, exposition naturelle ou vaccinale antérieure)
Pour interpréter l’implication d’un agent infectieux, il est préférable de mettre en évidence une séroconversion en réalisant une 1ère série de prélèvements en début d’infection (congénères en hyperthermie) et une 2ème série sur les mêmes animaux, trois semaines après (16).
Un des intérêts de la sérologie est aussi de pouvoir investiguer l’exposition des animaux vis-à-vis d’agents recherchés lors d’intervention tardive par rapport à l’épisode.
Dans ce cas, il est intéressant de prélever environ trois semaines après l’épisode 6 animaux exposés répondant aux critères suivants :
- les animaux exposés les plus âgés : au moins trois mois d’âge, mais de préférence de plus de six mois, pour limiter les titres élevés en anticorps maternels (alternative possible en allaitant : prélever les mères exposées à côté de leurs veaux),
- absence d’injection vaccinale sinon l’interprétation ne pourra porter que sur les valences concernées ou devra être prudente* si la dernière injection vaccinale remonte à moins de six mois.
Si ces conditions sont respectées, la mise en évidence de titres en anticorps spécifiques élevés (≥ +++ pour le kit BIOX K369 de Bio-X diagnostics®) sur la majorité des animaux est évocatrice d’une circulation récente des agents concernés.
La sérologie peut être également utilisée après une période ou saison à risque sur plusieurs animaux sentinelles > 6 mois d’âge et non vaccinés vis-à-vis des agents investigués* pour évaluer un statut du troupeau vis-à-vis de certains agents, sensibiliser les éleveurs et adapter le protocole de prévention.
Plusieurs propositions d’investigations sérologiques sont proposées (voir V).
*NB : Comment interpréter les résultats de l’ELISA pour des animaux vaccinés ?
Le risque de détection croisée des anticorps vaccinaux vis-à-vis des valences virales des vaccins injectable est élevé. Il est pour autant important de préciser qu’en dehors de validation spécifique, les tests ELISA commerciaux permettent rarement d’évaluer correctement une réponsevaccinale : ils ne sont pas spécifiques des souches ou des antigènes vaccinaux, ne ciblent pas particulièrement les anticorps neutralisants et n’évaluent pas la réponse cellulaire induite par des vaccins vivants atténués et certains vaccins inactivés adjuvés (17).
Par ailleurs, l’administration intranasale d’un virus vivant atténué non adjuvé peut induire une réponse mémoire systémique sans circulation d’anticorps au niveau sérique.
Aussi après une primo-infection naturelle ou une vaccination injectable, la demi-vie plasmatique des anticorps des agents respiratoires est relativement courte sans réexposition (18,19). Lors de nouvelle exposition naturelle ou vaccinale jusqu’à 1 an après, des anticorps peuvent être rapidement remis en circulation (17, 20).
Pour les agents bactériens, le risque de réponse croisée doit également être pris en compte et dépend des kits et des vaccins utilisés.
Par exemple, le principal kit multi-agents déployé en France (BioX K369) est basé sur un antigène LPS de Mannheimia haemolytica et ne détecte normalement pas d’anticorps induits par un vaccin dirigé contre les protéines IRP (17) ou la leucotoxine. Dans ce cas, sous réserve de vérification auprès du fabriquant de vaccin, l’interprétation sur cette valence reste donc possible malgré la vaccination.
Conclusion : proposition de schémas d’investigations et d’interprétations
Il ne peut pas y avoir de « recette » unique mais obligatoirement une combinaison de différents facteurs à prendre en compte pour améliorer son diagnostic étiologique :
- Recueil clinique, épidémiologique et des facteurs de risques,
- Maîtrise des conditions de prélèvement : choix ciblé des animaux, constitution de mélanges, moment du prélèvement, choix de la matrice, etc…
- Si mortalité : autopsie et histologie de manière systématique
- Diagnostic direct si on est en cours d’épisode, indirect si l’intervention est plus tardive,
- Connaissance des intérêts et limites des méthodes,
La conclusion sur l’implication ou non d’un agent se fera en fonction de la combinaison des différents résultats obtenus et implique de prendre en compte les facteurs d’influence recueillis, les caractéristiques propres à chaque agent infectieux recherché (portage au niveau respiratoire, potentiel pathogène primaire/secondaire, de virulence et de diffusion) et l’immunité des animaux concernés (exposition antérieure, immunité maternelle, vaccination).
Les propositions de démarches simplifiées d’intervention en termes de diagnostic présentées ici ne se veulent pas exhaustives : en effet, le recensement de l’ensemble des cas auxquels le praticien peut être confronté n’est pas possible. Ces propositions pourraient constituer pour lui une « boîte à outils » que seule son expertise, le recueil des différentes données qu’il pourra effectuer et sa connaissance de l’élevage pourront valoriser. Plusieurs propositions d’investigations et d’interprétations associant analyses directes et/ou indirectes sont proposées en présence d’épisode respiratoire, en amont d’une période à risque (avant les 1ers cas) ou après une saison à risque (schémas 1 à 4).
Bibliographie
1. PARDON B., BUCZINSKI S. Bovine Respiratory Disease Diagnosis: What Progress Has Been Made in Infectious Diagnosis? Vet Clin North Am Food Anim Pract. 2020; 36(2):425-444.
2. PARDON B., CALLENS J., MARIS J. ALLAIS L., VAN PRAET W., DEPREZ P., RIBBENS S. Pathogen-specific risk factors in acute outbreaks of respiratory disease in calves. J Dairy Sci. 2020; 103(3):2556-2566.
3. VAN DRIESSCHE L., VALGAEREN B., R., GILLE L., BOYEN F., DUCATELLE R., HAESEBROUCK F., PARDON B. A deep nasopharyngeal swab versus nonendoscopic bronchoalveolar lavage for isolation of bacterial pathogens from preweaned calves with respiratory disease. Journal of veterinary internal medicine. 2017; 31(3):946-953.
4. THOMAS A.C., BAILEY M., LEE M. MEAD A., MORALES-AZA B., REYNOLDS R., VIPOND B., FINN A., EISLER M.C. Insights into Pasteurellaceae carriage dynamics in the nasal passages of healthy beef calves. Sci Rep. 2019; 9(1):11943.
5. ANGEN O., THOMSEN J., LARSEN L.E. Respiratory disease in calves: microbiological investigations on trans-tracheally aspirated bronchoalveolar fluid and acute phase protein response. Vet Microbiol. 2009; 137(1-2):165-171
6. TIMSIT E., HALLEWELL J., BOOKER C., TISON N., AMAT S., ALEXANDER T.W. Prevalence and antimicrobial susceptibility of Mannheimia haemolytica, Pasteurella multocida, and Histophilus somni isolated from the lower respiratory tract of healthy feedlot cattle and those diagnosed with bovine respiratory disease. Vet Microbiol. 2017; 208:118-125.
7. LOWIE T., CALLENS J., MARIS J., RIBBENS S., PARDON B. Decision tree analysis for pathogen identification based on circumstantial factors in outbreaks of bovine respiratory disease in calves. Prev Vet Med. 2021; 196:105469.
8. TIMSIT E., CHRISTENSEN H., BAREILLE N., SEEGERS H., BISGAARD M., ASSIE S. Transmission dynamics of Mannheimia haemolytica in newly-received beef bulls at fattening operations. Vet Microbiol. 2013; 161(3-4):295-304.
9. TIMSIT E., ARCANGIOLI M.A., BAREILLE N., SEEGERS H., ASSIE S. Transmission dynamics of Mycoplasma bovis in newly received beef bulls at fattening operations. J Vet Diagn Invest. 2012; 24(6):1172-1176.
10. MEYER G., PELLETIER C., HERMAN N., DUCATEZ M., CASSARD H., SALEM E. Vers une identification de nouveaux virus respiratoires bovins. Nouveau Praticien Vétérinaire. 2015 ; 31(8):8-14.
11. JOZAN T., MELINE B. Vacciner les mères et les jeunes veaux dès leur plus jeune âge. L’exemple de la prévention des maladies respiratoires. Proceeding JNGTV. 2018 ; 655-660.
12. TIMSIT E., LE DREAN E., MAINGOURD C., BELLOC C., GUATTEO R., BAREILLE N., SEEGERS H., DOUART A., SELLAL E., ASSIE S. Detection by real-time RT-PCR of a bovine respiratory syncytial virus vaccine in calves vaccinated intranasally. Vet Rec. 2009; 165(8):230-233.
13. KLOMPMAKER A.F., BRYDENSHOLT M., MICHELSEN A.M., et al. Estimating Clinically Relevant Cut-Off Values for a High-Throughput Quantitative Real-Time PCR Detecting Bacterial Respiratory Pathogens in Cattle. Front Vet Sci. 2021; 8:674771.
14. Guide pratique de diagnostic. 2ème édition juillet 2015 réalisé par INP Toulouse et MSD Santé Animale.
15. PANCIERA R.J., CONFER A.W. Pathogenesis and pathology of bovine pneumonia. Vet Clin North Am Food Anim Pract. 2010;26(2):191-214.
16. FULTON R.W., CONFER A.W. Laboratory test descriptions for bovine respiratory disease diagnosis and their strengths and weaknesses: gold standards for diagnosis, do they exist ?. Can Vet J. 2012; 53(7):754-761.
17. BERGE A. C., JOZAN T., LEVESQUE C., VERTENTEN G. A Field Study Evaluating Humoral Immunity in Calves Vaccinated with Multivalent Bovine Respiratory Pathogen Vaccines. Folia Veterinaria. 2021; 65(4):20-30.
18. KIMMAN T. G., WESTENRINK F., SCHREUDER B. E., STRAVER P.J. Local and systemic antibody response to bovine respiratory syncytial virus infection and reinfection in calves with and without maternal antibodies. J. Clin. Microbiol. 1987; 25:1097-1106.
19. KIMMAN T. G., WESTENBRINK F., STRAVER P. J. Priming for local and systemic antibody memory responses to bovine respiratory syncytial virus : effect of amount of virus, virus replication, route of administration and maternal antibodies. Vet. Immunol. Immunopathol. 1989; 22:145—160.
20. MAKOSCHEY B., BERGE A. C. Review on bovine respiratory syncytial virus and bovine parainfluenza—usual suspects in bovine respiratory disease—A narrative review. BMC Vet. Res. 2021; 17:261—279.
21. LOY J., CLAWSON M. Rapid typing of Mannheimia haemolytica major genotypes 1 and 2 using MALDI-TOF mass spectrometry. Journal of Microbiological Methods.2017;136:30-33
22. KLIMA C., ZAHEER R., BRIGGS R., McALLISTER T. A multiplex PCR assay for molecular capsular serotyping of Mannheimia haemolytica serotypes 1, 2, and 6. Journal of Microbiological Methods.2017; 139 :155-160.
23. COZENS D., SUTHERLAND E., LAUDER M., TAYLOR G. BERRY C., DAVIES R. Pathogenic Mannheimia haemolytica invades differentiated bovine airway epithelial cells. Infection and Immunity. 2019; 87(6): e00078-19.
24. NAKAY I., NAKAZAWA M., HASHIMOT Y., IKEUCHI T., NOMA S., TORIKAI Y., EZAKI T. Isolation and characterization of Pasteurella trehalosi from cattle. Journal of the Japan Veterinary Medical Association. 1995; 48:750–754.
25. COLLINS RL. Bibersteinia trehalosi in cattle – another component of the bovine respiratory disease complex? Cattle Practice.2011;19:9–12.
26. BROWN S., BYCROFT K., ADAM K., COLLETT M. Acute fibrinous pleuropneumonia and septicaemia caused by Bibersteinia trehalosi in neonatal calves in New Zealand. New Zealand Veterinary Journal. 2021; 69(1):51-57.
27. CORTESE V., BRAUN D., CROUCH D., TOWNSEND C., ZUKOWSKI B. Case report – peracute to acute fatal pneumonia in cattle caused by Bibersteinia trehalosi. Bovine Practitioner. 2012;46,138–42.
28. BOWERSOCK T., SOBECKI B., TERRILL S., MARTINON N., MEINERT T., LEYH R. Efficacy of a multivalent modified live virus vaccine containing a Mannheimia haemolytica toxoid in calves challenge exposed with Bibersteinia trehalosi. American Journal of Veterinary Research. 2014; 75:770-776.
29. VAN DRIESSCHE L., VANNESTE K., BOGAERTS B., DE KEEESMAECKER S., Nancy H. ROOSENS N., HAESEBROUCK F., DE CREMER L., DEPREZ P., PARDON B., BOYEN F. Isolation of Drug-Resistant Gallibacterium anatis from Calves with Unresponsive Bronchopneumonia, Belgium. Emerging Infectious Diseases. 2020; 26:721-730.
GP-FR-NON-220400025

